分子动力学(九)
分子动力学模拟的主要步骤
三、方均根偏差
方均根偏差(root mean square deviation, RMSD)反映了任意时刻的体系构象与初始构象之间的偏差。RMSD的大小反映了体系的总体柔性与稳定性。RMSD值越小,表明模拟体系总体越稳定。具体的计算操作为:
- 输入命令:$ ptraj ../../complex_vac.prmtop rmsd.in。图1给出了rmsd.in文件的内容。其中,第1行为输入轨迹;第2行表示镜像范围;第3行为计算体系总体Cα原子的RMSD,时间间隔为1.0 ps。
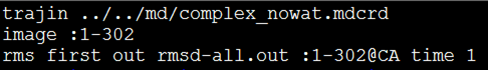
图1 文件rmsd.in的内容
四、方均根涨落
方均根涨落(root mean square fluctuation, RMSF)可用来反映体系在整个模拟过程中相对于平均结构所发生的构象变化。较大的RMSF值说明对应的残基柔性较大。下面给出了计算RMSF的方法:
输入命令:$ ptraj ../../complex_vac.prmtop rmsf.in。图2给出了rmsf.in文件的内容,其中第1行表示输入轨迹;第2行表示镜像残基范围;第3行表示计算体系总体Cα原子的RMSF值。
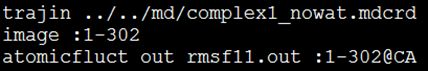
图2文件rmsf.in的内容
五、两点间距离、角度及二面角
原子间的距离、角度和二面角随模拟时间的变化最能直观描述原子间的相互作用。如:判断两个残基之间是否具有较为显著的相互作用,可以计算两个残基质心之间的距离,并分析其随模拟时间的变化;分析氢键时,常需计算氢键中氢供体原子-氢原子-氢受体原子形成的角度;4个原子所构成的二面角一般用作分析柔性较大的残基。具体的计算方法为:
- 输入命令:$ ptraj ../../complex_vac.prmtop all.in > all.out。图3给出了文件all.in的内容,其中第1行表示输入轨迹;第2行表示镜像范围;第3行为计算279号残基的Cα原子与302号小分子6JS1003中C23原子间的距离,输出文件为a.out;第4行表示计算94号残基氧原子、6JS1003的H4原子以及6JS1003的N7原子构成的角度;第5行表示计算残基130~140段质心、残基179~184段质心、残基229~250段质心及残基279~300段质心所构成的二面角。
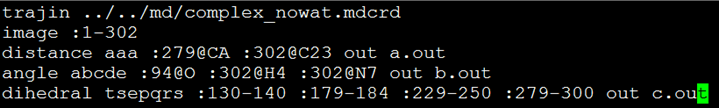
图3 文件all.in的内容
这里还对一些其他分析进行简单得介绍,具体得分析过程会在后续给出。
六、能量分解
用MM/GBSA(molecular mechanics/generalized Born surface area)方法进行能量分解分析。其基本思想是把每个残基的能量贡献近似分为力学(molecule mechanics, MM)方法计算的真空分子内能、用广义波恩(generalized Born, GB)模型计算的极性溶剂化能,以及用LCPO算法计算的非极性溶剂化能,并且把能量分解到残基的主链原子核侧链原子上。这里,真空分子内能可分为极性的静电相互作用和非极性的范德华相互作用两部分。LCPO算法认为非极性溶剂化能与溶剂可接近表面积(solvent accessible surface area, SASA)呈正相关。通过MM/GBSA能量分解,可以观察到蛋白中的主要残基对小分子结合所做出的贡献。
七、自由能曲面
自由能曲面(free energy landscape, FEL)是通过研究自由能曲面图的最小值(即体系最稳定的状态)和分界点(即体系变化过程中的一个短暂的状态)来表示生物学过程中发生的动力学变化的一种分析方法。一些主成分(principal component, PC)可以用以表示重要的动力学过程,这些PC也可以来绘制自由能曲面图。
八、聚类分析
使用MMTSB工具,对MD模拟得到的体系构象进行聚类分析(cluster analysis)。分析过程依据Daura等提出的观点:逐一计算各构象之间的RMSD值,以此为基础建立RMSD矩阵(N×N, N为构象数)。人为设定一个RMSD阈值,在任意两构象RMSD值之间进行比较,若其差值小于此阈值则归为一类。以每一类中能量最低的构象作为该类的代表构象。