ProDy的使用—— Druggability的计算
在ProDy的众多功能中,Druggability是一项十分重要并且有意义的,也可称为“类药性口袋”计算,是在蛋白质靶点的研究中一个非常重要的概念。Druggability是指在蛋白质靶点表面存在的特异性的结合位点的位置。目前大量蛋白质的晶体结构被解析出来,然而由于技术上或性质上的问题,导致不能清晰地认识每个蛋白质的活性位点的具体位置,而Druggability的出现正好可以解决这个问题,这对于人们更加深入认识蛋白质的结合和功能具有重要的意义。
Druggability是基于VMD中的GUI界面以及python模块的对模拟轨迹的分析套件。其中心思想是采用小分子探针,主要是异丙醇、乙酰胺、乙酸、异丙胺和异丁烷,与蛋白质靶点进行长时间的分子动力学模拟,并计算每个探针分子与蛋白质的结合自由能,进而评价靶点表面最有可能的结合位点的位置。其中,探针分子的结合自由能可以采用一下的公式计算得到:
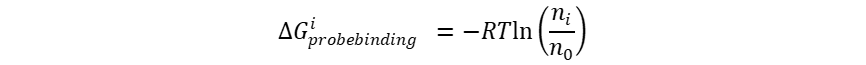
在这里, 表示探针密度与期望密度的比值,R表示气体常数,T表示绝度温度。下面我们将以PFV整合酶的晶体结构3S3M为例,对Druggability的计算过程进行简单的介绍。
为了计算的稳定性以及结果的可靠性,本例中的所有计算过程均在Linux系统下完成,需要准备的软件有VMD和NAMD,安装过程不再赘述。
- 准备文件:事先从RCSB网站上下载3S3M的晶体结构数据,并用VMD打开另存为PSF格式的文件
- 在Linux下打开VMD的可视化界面,点击Extensions-Modeling-DruGUI,即出现Druggability的计算界面,在“Prepare System”中点击选择准备好的PSF和PDB文件,并在Output Options中选择Output folder的位置,以及Output prefix,在探针分子组成(Probe composition)中可以根据不同需要选择探针分子的组成及比例,模拟的长度(Sim length)可以设置大一点,有利于提高结果的准确性,最后点击Prepare System
- 完成体系准备工作后,在输出文件目录会出现3s3m_min和3s3m_sim两个文件夹,在Linux系统下用NAMD依次执行3s3m_min文件夹中的min.conf,以及3s3m_sim 文件夹中的eq1.conf、eq2.conf、eq3.conf和sim.conf,具体计算的命令为“namd2 min.conf > min.log”,运算过程会比较耗时,需要注意的是在计算过程中需要先执行优化过程(min.conf),并且模拟过程的文件必须按照顺序依次进行,运算结束后可以得到eq1.dcd,eq2.dcd,eq3.dcd和sim.dcd三个轨迹文件
- 模拟完成后,需要用VMD对模拟的轨迹进行分析,并计算得到探针格点(probe grid)。在DruGUI中选择Calculate Grids,并在DCDs中添加计算得到的模拟轨迹文件eq1.dcd,eq2.dcd,eq3.dcd和sim.dcd,其余参数根据需要进行修改,如图1所示
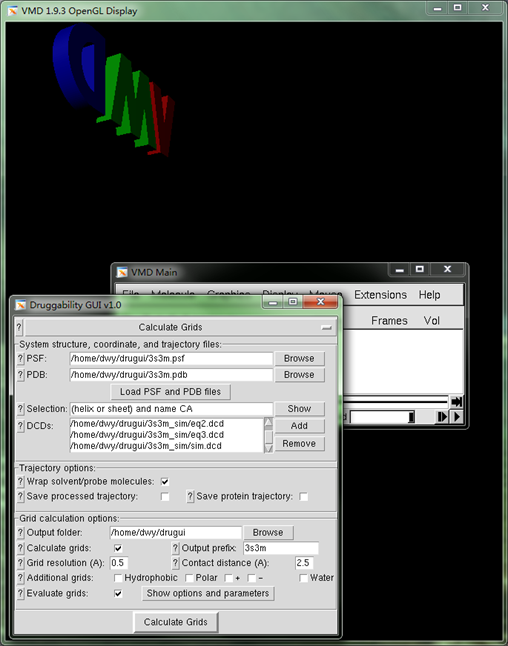
图1 计算探针格点
- 计算完成后,得到各种探针分子在蛋白质表面的分布情况,包括3s3m_ACAM.dx,3s3m_ACET.dx,3s3m_IPAM.dx,3s3m_IPRO.dx文件。最后采用Assess Druggability模块进行分析计算,在Probe grid files中添加所有.dx文件,其他参数根据需要进行修改,本例中采用默认参数,如图2所示,最后点击Assess Druggability完成计算
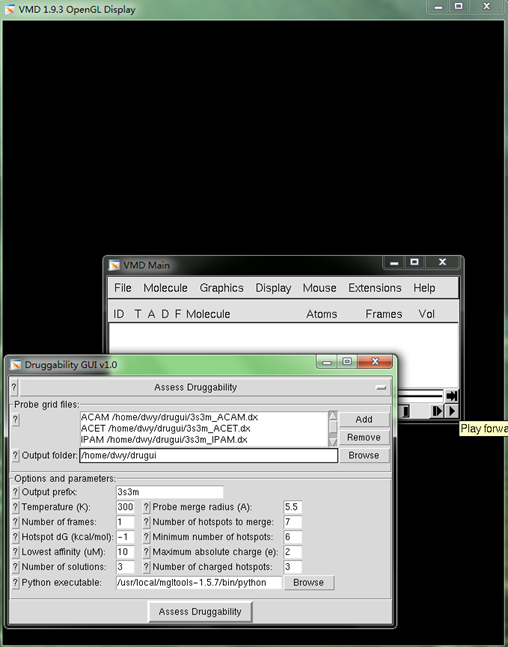
图2 计算Druggability
- 最终得到的3s3m_all_hotspots.pdb中即包含了所有探针分子的信息,并且VMD根据探针分子所在的区域将蛋白质表面自动划分为6个结合热点区,圆形即表示探针分子,不同颜色表示结合自由能的大小即结合的强弱
对上述探针分子-蛋白质结合图进行修饰可以得到如下更为直观的结合位点图(图3。在考虑了蛋白质和探针分子的溶剂效应和耦合效应之后,计算得到探针分子的结合自由能,图中红色表示结合自由能低的点(结合能力较强),蓝色表示结合自由能较高的点(结合能力较弱),白色则表示中等。观察发现3s3m蛋白质结构表面的探针分子中,只有图中所示的一处红色点,即该点是所有探针分子中结合能力最强的,它与其余4个点所占据的口袋位置正好是目前公认并且多篇文献中也有报道的抑制剂结合的活性位点。因此,ProDy软件中的Druggability模块的计算可信度较高,对于未知结合位点的蛋白质来讲是一个较好的借鉴。
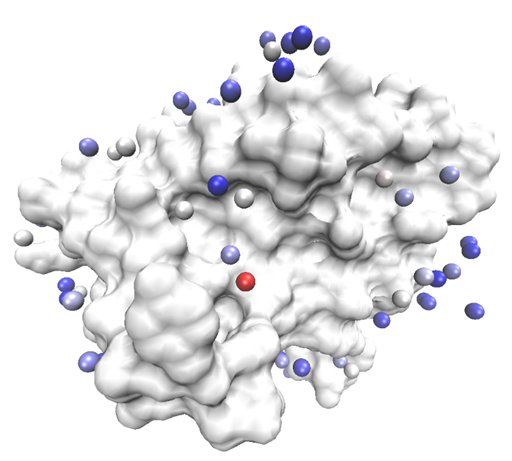
图3 探针分子在蛋白质表面的分布