PLS分析
- 鼠标选中生物活性数据列(pIC50)和CoMFA列;在电子表格中的菜单栏选择QSAR,并在下拉菜单中点击Partial Least Squares。
- 在弹出的对话框中,设置Components值为6;关闭Use SAMPLES;在Validation栏中勾选Leave-One-Out;勾选Column Filtering,并设为2.0;在Scaling项中选择CoMFA Standard;设置完成后,点击Do PLS,开始进行交叉验证。验证结果将存为以Analysis Name命名的*.pls文件,如:ALIGNRING1_1x.pls。
注意:(1)进行交叉验证时,Components的值可以根据实际需要修改,一般情况下设为6即可得到最佳模型;(2)Column Filtering可以自动过滤与因变量相关性不大的描述符,其值越大,系统过滤的描述符越多,计算量越小,计算速度越快。因此可在保证结果准确性的前提下,设置较大的Column Filtering值,以节约计算资源。
- 对CoMSIA模型进行PLS分析时,操作类似。在点击QSAR前,选中生物活性值(pIC50)与全部5中分子场数值即可。
点击Do PLS后,PLS的窗口并不会消失。在SYBYL主界面左下角的命令窗口中,可以观察到交叉验证后的结果。交叉验证得到的最佳主成分数为6,交叉验证系数R2(q2)大于0.5较为合理。
- 返回PLS对话框,更改Components值为交叉验证后得到最佳主成分数,由于本例中为6,故不需要更改;在Validation栏中勾选No Validation,关闭Column Filtering;点击Do PLS,进行常规回归分析。分析结果也将以*.pls的形式保存,如:ALIGNRING1_2xx.pls。
这时,可在命令窗口查看CoMFA模型的拟合结果。当模型的相关系数R2大于0.9,F值越大越好,即可证明建立的CoMFA模型具有较好的预测能力,和较为显著的统计学意义。
- 在PLS对话框中点击End关闭对话框,完成模型的建立。
CoMSIA的操作与CoMFA类似,具体操作过程此处不再赘述。
等势图的生成
- 鼠标点击菜单栏中的QSAR,在下拉菜单中选择View QSAR。点击CoMFA即弹出CoMFA等势图参数设置对话框;点击CoMSIA则弹出CoMSIA的对话框。
CoMFA和CoMSIA的对话框中的选项略有不同。其中相同之处在于:
- 在对话框的TYPE OF FIELD TO DISPLAY栏中勾选PLS Analysis,选择常规回归分析保存的文件,StDev*Coeff表示。
- 在CONTOUR SPECIFICATIONS栏中,选择Contour by: Contribution,Display as: Solid。
- 每种分子场在没有特殊要求的情况下,Favored截断值均设为80.0,Disfavored截断值均设为20.0。在CoMFA模型中,只有两种分子场,即Sterics和Electrostatics,表示每种分子场的色块颜色不可更改(黄色和绿色表示立体场,红色和蓝色表示静电场);而在CoMSIA模型中则有5中分子场,表示分子场的颜色可以更改。根据做图需要,勾选相应的分子场后即可在操作界面上显示,可以单独做图,也可合并做图。如:
- 勾选Show Sterics (Bulk) in D2和Save to File (s);去掉Show Electrostatics (H+) in D3和Examine Predicted vs Actual in D1前的勾,;点击Show and Quit,在操作界面上出现立体场的色块分布。
- 在电子表格中,选中模板分子所在行;在行头序号处点击鼠标右键,在下拉菜单中点击Put Structures in SYBYL-X Window,即可将该分子添加到色块中。
按照类似的操作,可以得到静电场的等势图。
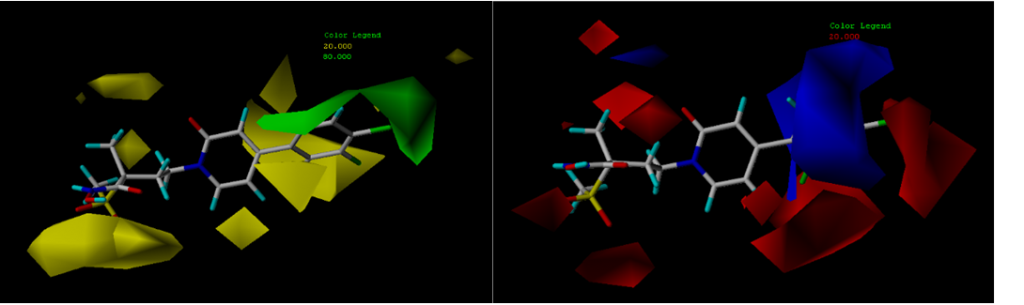
图1CoMFA等势图
- 绘制CoMSIA疏水场和氢键场等势图时,分别勾选Hydrophobic和Donor、Acceptor,并根据制图需要调整色块的颜色;勾选 Save to File (s);点击Show and Quit。
操作界面中显示色块后。利用同样的操作将模板分子显示在色块中,即完成了CoMSIA等势图。
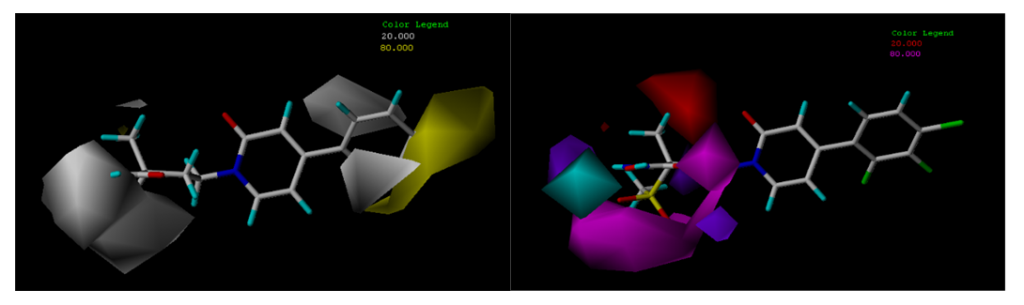
图2 CoMSIA等势图
生物活性值的预测
- 选中电子表格的空列,点击鼠标右键,在下拉菜单中选择Add A Computed Column,在弹出的对话框中选择PREDICT,点击OK;选择用于预测的电子表格,如:123.tbl,点击OK。
- 选择用于计算预测值的模型,输入名称,点击OK,系统将会自动计算并将预测结果填入表格中。
需要注意的是:(1)在选择用于计算预测值的模型时,无论是CoMFA还是CoMSIA,不能选择交叉验证时的模型,而要使用常规回归分析的模型;(2)在对测试集进行预测时,必须将记录训练集信息的电子表格打开,否则将无法进行预测。
得到预测值后,可将其与生物活性值一并拷入制图软件(如:Origin等)中制图。