基于配体的药物分子设计(五)
CoMFA和CoMSIA作为3D-QSAR的主要研究方法,已作为SYBYL、Discovery Studio等大型分子模拟软件中的一个模块实现了商业化,被广泛应用于药学、化学等各学科领域。这里以构建3D-QSAR方程的化合物为例,主要介绍SYBYL-X中CoMFA和CoMSIA的具体操作。
1. 数据库的建立
- 此步骤与2D-QSAR的操作一致,详细操作请参看前文。
2. 分子叠合
分子叠合的好坏是影响模型质量的一个十分重要的因素。在SYBYL中提供了多种叠合的策略,如按照公共骨架叠合,按照药效团模型叠合,按照分子对接结果叠合等。为了得到优秀的CoMFA模型,可能需要尝试多种叠合方式。下面以按照公共骨架叠合为例,介绍具体操作:
- 鼠标点击菜单栏中的File,在下拉菜单中选择Database,点击Align Database。
- 在弹出的对话框中Database to Align一栏中选择用于叠合的数据库文件,如MSH.mdb;然后在Template Molecule一栏中选择数据库中的一个分子作为模板分子,值得注意的是:在CoMFA中假定所有分子均以相同活性方式结合于同一位点,故常选取活性最高化合物为模板分子。
在Common Substructure栏中输入用于叠合的公共骨架。鼠标左键+Shift在模板分子上进行选择或在弹出的Atom Expression对话框进行勾选。选择结束后点击OK,操作界面中模板分子的非选择部分将会隐去。
选择时须注意:(1)骨架必须是数据库中所有化合物所共有;如果不是,不含有该骨架的分子将无法自动叠合;(2)选择的骨架结构中不能包含氢原子。
- Grid Orientation项选择Inertial,Put Info项选择New Database,Align项选择All Molecules。确认无误后点击Apply开始进行分子叠合。
- 分子叠合过程中如果遇到不含有公共结构的分子,系统会弹出对话框提示。点击确认后,系统将继续叠合。叠合完成后,在弹出的对话框中输入新数据库的名称,点击OK即可。
注意:(1)在实际QSAR研究中,除了用于构建模型的分子外,还需选择一些分子组成测试集,用于模型预测能力的检验。测试集中的分子也需要进行叠合,且模板分子须与训练集叠合时选择的模板分子一致;(2)为了使CoMFA力场中的信息能被最大效率的利用,提高CoMFA模型的交叉验证系数以及其他科研工作者更好地重复这一工作,需要冻结模板分子的构象。
- 点击菜单栏中的View,在下拉菜单中选择Transformation,点击Freeze All即可。
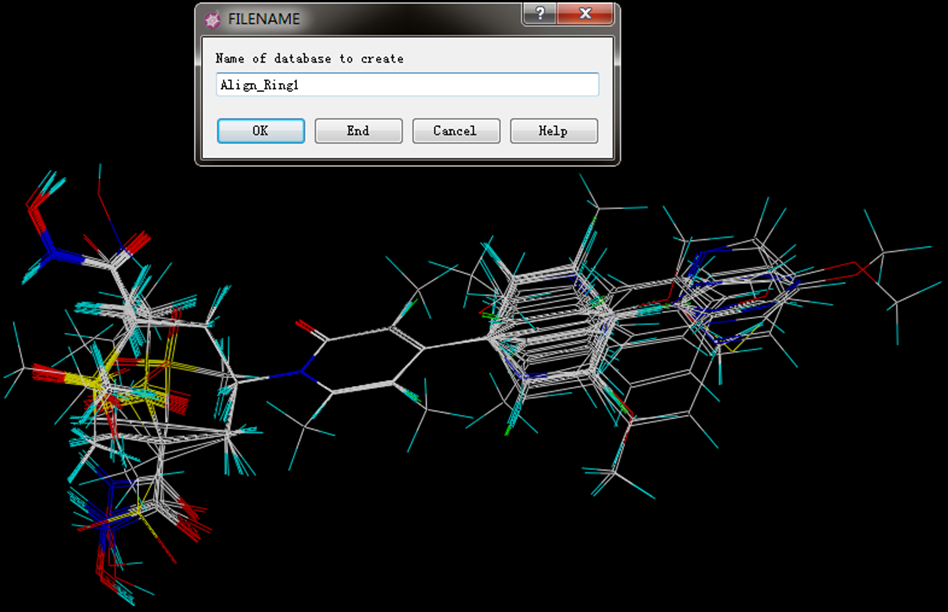
图1 分子叠合结果
3. 电子表格的生成
此步骤与2D-QSAR中的操作相似。不同之处在于,选择生成表格的数据库文件时应使用分子叠合后新生成的数据库文件。
生成的表格中不含有生物活性数据,只有化合物的名称和结构式,需要手动添加。添加的数据形式应为负对数形式,如:pIC50。
- 在空表头处,点击鼠标右键,在弹出的菜单中点击Add Empty Column;在弹出的对话框中输入数据的名称,数据类型选择Number,点击OK。之后可将生物活性数据复制粘贴至相应位置。
- 数据量较大时,也可以先将拟添加的数据按照电子表格中的形式保存为文本文档(*.txt),再在Spreadsheet的工具栏中点击Import File导入。
4. 添加格点
- 在空白表头处点击鼠标右键,在菜单中选择Add A Computed Column。如果建立CoMFA模型,则在弹出的对话框中选择COMFA,点击OK;如果建立CoMSIA模型,则选择COMSIA。
- 建立CoMFA模型需要在弹出的对话框中设定能量阈值,一般情况下立体场和静电场的阈值均设为30.0 kcal/mol。其他参数没有特殊要求,均为缺省值。点击Add Column,计算并添加格点。
- 建立CoMSIA模型需要在弹出的对话框中选择分子场类型,一般选择单独的5种场。衰减因子一般设为0.3。点击Add Column开始添加格点。
两种模型在计算完成后,都会弹出纵列描述对话框,直接点击OK;接着会弹出字符串对话框,提示输入数据,直接点击End。格点数据便会自动添加到电子表格中,数据填充完毕后,点击File,选择Save保存电子表格,如:123.tbl。